摘要:DNA的折疊和運動,以及某些蛋白質(zhì)的積累,取決于基因是活躍還是不活躍。
日本福岡——九州大學(xué)的研究人員揭示了DNA特定區(qū)域之間的空間距離是如何與基因活動的爆發(fā)聯(lián)系在一起的。利用先進的細(xì)胞成像技術(shù)和計算機建模,研究人員表明,DNA的折疊和運動,以及某些蛋白質(zhì)的積累,取決于基因是活躍還是不活躍。這項研究發(fā)表在12月6日的《科學(xué)進展》雜志上,揭示了基因表達(dá)的復(fù)雜世界,并可能為基因表達(dá)調(diào)節(jié)不當(dāng)引起的疾病帶來新的治療技術(shù)。
基因表達(dá)是發(fā)生在細(xì)胞內(nèi)的一個基本過程,主要有兩個階段:轉(zhuǎn)錄(DNA被復(fù)制成RNA)和翻譯(RNA被用來制造蛋白質(zhì))。為了讓每個細(xì)胞在體內(nèi)發(fā)揮其特定的功能,或者對不斷變化的環(huán)境做出反應(yīng),必須在適當(dāng)?shù)臅r間產(chǎn)生適量的蛋白質(zhì),這意味著必須小心地打開和關(guān)閉基因。
 圖1 轉(zhuǎn)錄爆發(fā)過程中基因組區(qū)域鄰近性的轉(zhuǎn)錄偶聯(lián)變化
圖1 轉(zhuǎn)錄爆發(fā)過程中基因組區(qū)域鄰近性的轉(zhuǎn)錄偶聯(lián)變化以前,基因轉(zhuǎn)錄被認(rèn)為是一個連續(xù)的、平穩(wěn)的過程。但隨著更好的技術(shù)來觀察單個細(xì)胞,科學(xué)家們現(xiàn)在知道轉(zhuǎn)錄發(fā)生在短時間內(nèi),不可預(yù)測的爆發(fā)。
“一個基因會隨機開啟幾分鐘,大量的RNA會產(chǎn)生。然后,基因會突然再次關(guān)閉,”來自九州大學(xué)生物調(diào)控醫(yī)學(xué)研究所的Hiroshi Ochiai教授說,他是該研究的資深作者。“它發(fā)生在幾乎所有基因和所有生物中,從植物到動物再到細(xì)菌。”
這種不穩(wěn)定和動態(tài)的轉(zhuǎn)錄性質(zhì),被稱為轉(zhuǎn)錄爆發(fā),是控制單個細(xì)胞中基因活性的關(guān)鍵機制。這就是為什么同一組織或培養(yǎng)環(huán)境中的細(xì)胞在基因表達(dá)水平上表現(xiàn)出差異的原因之一,這對早期胚胎發(fā)育和癌癥進化等過程至關(guān)重要。然而,爆炸背后的確切機制仍然未知。
在這項研究中,研究人員決定研究被稱為增強子和啟動子的DNA序列的作用,以及它們的空間距離如何影響轉(zhuǎn)錄爆發(fā)。啟動子通常位于基因旁邊,是執(zhí)行轉(zhuǎn)錄的蛋白質(zhì)附著在DNA上的地方。另一方面,增強子通常距離基因數(shù)十萬個堿基,但隨著DNA鏈的移動和折疊,增強子仍然可以在三維空間中靠近基因,從而放大基因活性。
Ochiai說:“我們相信增強子在轉(zhuǎn)錄在活動爆發(fā)中發(fā)生的原因中起著至關(guān)重要的作用,但到目前為止,研究還不清楚。”
為了驗證這個想法,Ochiai和他的團隊使用了一種稱為seq-DNA/RNA-IF-FISH的先進成像技術(shù),該技術(shù)可以用熒光探針標(biāo)記DNA, RNA和特定蛋白質(zhì)。這種三層技術(shù)允許研究人員同時在單個小鼠胚胎干細(xì)胞的三維空間中捕獲DNA、RNA和特定蛋白質(zhì)的位置。有了這些信息,研究小組可以確定某些基因是開啟還是關(guān)閉,看到啟動子和增強子在活動爆發(fā)時如何相互作用,以及蛋白質(zhì)在哪里積聚,以前所未有的細(xì)節(jié)水平。
作為一個例子,研究人員專注于一種名為Nanog的基因,這是6號染色體上長度為77萬個堿基的DNA,它有一個啟動子和三個增強子區(qū)域,已知在培養(yǎng)的小鼠胚胎干細(xì)胞中會發(fā)生轉(zhuǎn)錄破裂。
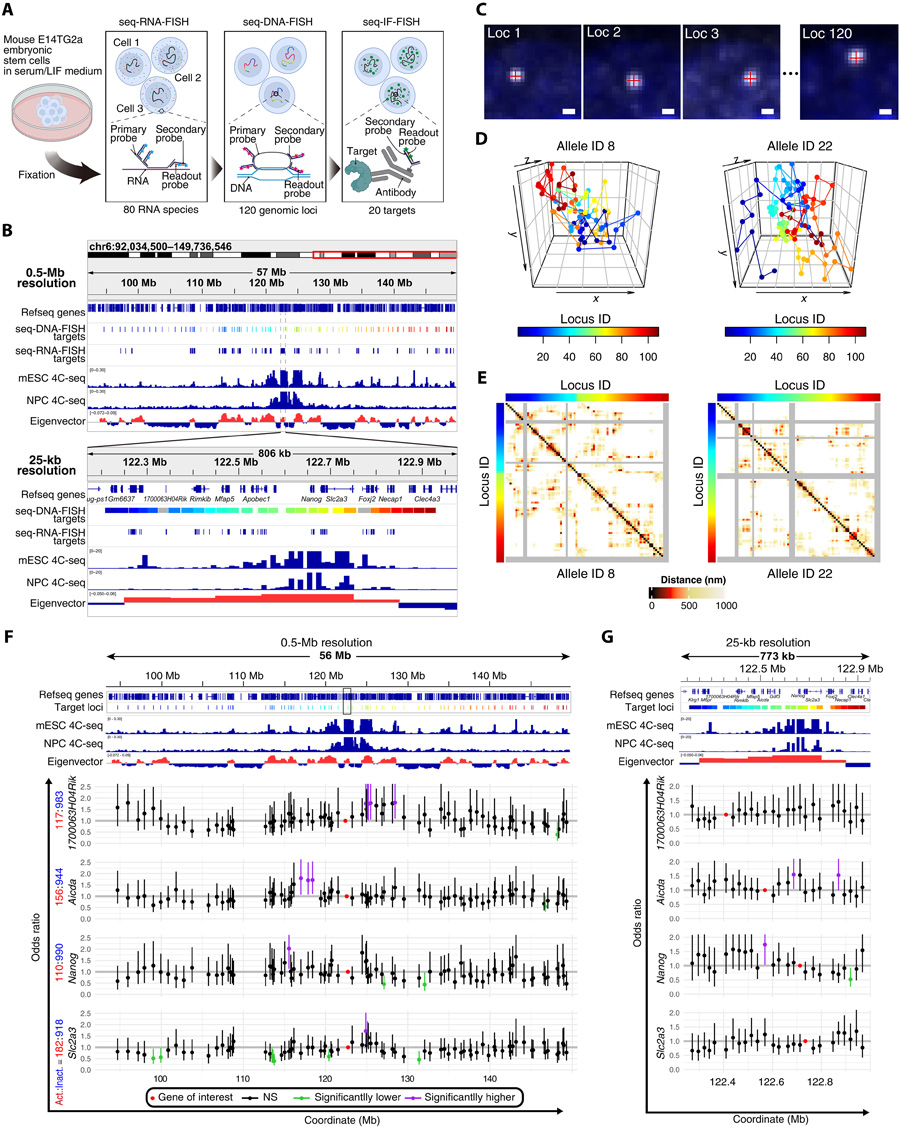
圖2 轉(zhuǎn)錄活性狀態(tài)特異性基因組鄰近性
研究人員發(fā)現(xiàn),在Nanog RNA存在的成像細(xì)胞中(這意味著該基因是活躍的),距離最遠(yuǎn)的增強子位于Nanog基因附近的空間。相反,當(dāng)Nanog不活躍時,成像顯示相同的增強子區(qū)域在物理上更遠(yuǎn)。
此外,科學(xué)家們還發(fā)現(xiàn),當(dāng)Nanog活躍時,參與調(diào)節(jié)轉(zhuǎn)錄的蛋白質(zhì)也在增強子和啟動子周圍的區(qū)域積累。
為了更好地理解這一機制,Ochiai和他的團隊使用計算機建模來模擬DNA的不同部分是如何在細(xì)胞內(nèi)相互作用和移動的,無論Nanog基因是活躍的還是不活躍的。
他們通過使用成像實驗的數(shù)據(jù)來開發(fā)他們的模型,以繪制DNA不同區(qū)域相互作用的頻率以及DNA在空間中如何折疊的“地圖”。利用這張地圖,該模型隨后模擬了DNA鏈如何隨機移動。
該模型預(yù)測,當(dāng)處于激活狀態(tài)時,每個增強子區(qū)域與啟動子相互作用的時間是基因不活躍時的兩倍多。
該模型顯示,這些較長時間的相互作用是由于DNA周圍的“摩擦”而發(fā)生的。當(dāng)Nanog活躍時,由于蛋白質(zhì)和RNA的積累,液體變得更加粘稠,導(dǎo)致模擬的DNA鏈移動緩慢。因此,這種基因能夠在更長的時間內(nèi)保持活躍。相比之下,當(dāng)Nanog不活躍時,模擬DNA移動得更快,這意味著啟動子和增強子沒有時間相互作用。
“模型表明,由于這些強化循環(huán),爆炸是穩(wěn)定的,”O(jiān)chiai總結(jié)道。“當(dāng)然,這只是一個模擬。下一步是證明這種機制也發(fā)生在細(xì)胞中。”
參考資料
[1] Transcription-coupled changes in genomic region proximities during transcriptional bursting
摘要:DNA的折疊和運動,以及某些蛋白質(zhì)的積累,取決于基因是活躍還是不活躍。
日本福岡——九州大學(xué)的研究人員揭示了DNA特定區(qū)域之間的空間距離是如何與基因活動的爆發(fā)聯(lián)系在一起的。利用先進的細(xì)胞成像技術(shù)和計算機建模,研究人員表明,DNA的折疊和運動,以及某些蛋白質(zhì)的積累,取決于基因是活躍還是不活躍。這項研究發(fā)表在12月6日的《科學(xué)進展》雜志上,揭示了基因表達(dá)的復(fù)雜世界,并可能為基因表達(dá)調(diào)節(jié)不當(dāng)引起的疾病帶來新的治療技術(shù)。
基因表達(dá)是發(fā)生在細(xì)胞內(nèi)的一個基本過程,主要有兩個階段:轉(zhuǎn)錄(DNA被復(fù)制成RNA)和翻譯(RNA被用來制造蛋白質(zhì))。為了讓每個細(xì)胞在體內(nèi)發(fā)揮其特定的功能,或者對不斷變化的環(huán)境做出反應(yīng),必須在適當(dāng)?shù)臅r間產(chǎn)生適量的蛋白質(zhì),這意味著必須小心地打開和關(guān)閉基因。
 圖1 轉(zhuǎn)錄爆發(fā)過程中基因組區(qū)域鄰近性的轉(zhuǎn)錄偶聯(lián)變化
圖1 轉(zhuǎn)錄爆發(fā)過程中基因組區(qū)域鄰近性的轉(zhuǎn)錄偶聯(lián)變化以前,基因轉(zhuǎn)錄被認(rèn)為是一個連續(xù)的、平穩(wěn)的過程。但隨著更好的技術(shù)來觀察單個細(xì)胞,科學(xué)家們現(xiàn)在知道轉(zhuǎn)錄發(fā)生在短時間內(nèi),不可預(yù)測的爆發(fā)。
“一個基因會隨機開啟幾分鐘,大量的RNA會產(chǎn)生。然后,基因會突然再次關(guān)閉,”來自九州大學(xué)生物調(diào)控醫(yī)學(xué)研究所的Hiroshi Ochiai教授說,他是該研究的資深作者。“它發(fā)生在幾乎所有基因和所有生物中,從植物到動物再到細(xì)菌。”
這種不穩(wěn)定和動態(tài)的轉(zhuǎn)錄性質(zhì),被稱為轉(zhuǎn)錄爆發(fā),是控制單個細(xì)胞中基因活性的關(guān)鍵機制。這就是為什么同一組織或培養(yǎng)環(huán)境中的細(xì)胞在基因表達(dá)水平上表現(xiàn)出差異的原因之一,這對早期胚胎發(fā)育和癌癥進化等過程至關(guān)重要。然而,爆炸背后的確切機制仍然未知。
在這項研究中,研究人員決定研究被稱為增強子和啟動子的DNA序列的作用,以及它們的空間距離如何影響轉(zhuǎn)錄爆發(fā)。啟動子通常位于基因旁邊,是執(zhí)行轉(zhuǎn)錄的蛋白質(zhì)附著在DNA上的地方。另一方面,增強子通常距離基因數(shù)十萬個堿基,但隨著DNA鏈的移動和折疊,增強子仍然可以在三維空間中靠近基因,從而放大基因活性。
Ochiai說:“我們相信增強子在轉(zhuǎn)錄在活動爆發(fā)中發(fā)生的原因中起著至關(guān)重要的作用,但到目前為止,研究還不清楚。”
為了驗證這個想法,Ochiai和他的團隊使用了一種稱為seq-DNA/RNA-IF-FISH的先進成像技術(shù),該技術(shù)可以用熒光探針標(biāo)記DNA, RNA和特定蛋白質(zhì)。這種三層技術(shù)允許研究人員同時在單個小鼠胚胎干細(xì)胞的三維空間中捕獲DNA、RNA和特定蛋白質(zhì)的位置。有了這些信息,研究小組可以確定某些基因是開啟還是關(guān)閉,看到啟動子和增強子在活動爆發(fā)時如何相互作用,以及蛋白質(zhì)在哪里積聚,以前所未有的細(xì)節(jié)水平。
作為一個例子,研究人員專注于一種名為Nanog的基因,這是6號染色體上長度為77萬個堿基的DNA,它有一個啟動子和三個增強子區(qū)域,已知在培養(yǎng)的小鼠胚胎干細(xì)胞中會發(fā)生轉(zhuǎn)錄破裂。

圖2 轉(zhuǎn)錄活性狀態(tài)特異性基因組鄰近性
研究人員發(fā)現(xiàn),在Nanog RNA存在的成像細(xì)胞中(這意味著該基因是活躍的),距離最遠(yuǎn)的增強子位于Nanog基因附近的空間。相反,當(dāng)Nanog不活躍時,成像顯示相同的增強子區(qū)域在物理上更遠(yuǎn)。
此外,科學(xué)家們還發(fā)現(xiàn),當(dāng)Nanog活躍時,參與調(diào)節(jié)轉(zhuǎn)錄的蛋白質(zhì)也在增強子和啟動子周圍的區(qū)域積累。
為了更好地理解這一機制,Ochiai和他的團隊使用計算機建模來模擬DNA的不同部分是如何在細(xì)胞內(nèi)相互作用和移動的,無論Nanog基因是活躍的還是不活躍的。
他們通過使用成像實驗的數(shù)據(jù)來開發(fā)他們的模型,以繪制DNA不同區(qū)域相互作用的頻率以及DNA在空間中如何折疊的“地圖”。利用這張地圖,該模型隨后模擬了DNA鏈如何隨機移動。
該模型預(yù)測,當(dāng)處于激活狀態(tài)時,每個增強子區(qū)域與啟動子相互作用的時間是基因不活躍時的兩倍多。
該模型顯示,這些較長時間的相互作用是由于DNA周圍的“摩擦”而發(fā)生的。當(dāng)Nanog活躍時,由于蛋白質(zhì)和RNA的積累,液體變得更加粘稠,導(dǎo)致模擬的DNA鏈移動緩慢。因此,這種基因能夠在更長的時間內(nèi)保持活躍。相比之下,當(dāng)Nanog不活躍時,模擬DNA移動得更快,這意味著啟動子和增強子沒有時間相互作用。
“模型表明,由于這些強化循環(huán),爆炸是穩(wěn)定的,”O(jiān)chiai總結(jié)道。“當(dāng)然,這只是一個模擬。下一步是證明這種機制也發(fā)生在細(xì)胞中。”
參考資料
[1] Transcription-coupled changes in genomic region proximities during transcriptional bursting



